Was sind Gene Drives?
Mit Hilfe von neuen Gentechnikverfahren wie CRISPR/Cas9 wurden in den letzten Jahren sogenannte Gene Drives entwickelt, mit denen der Mensch neue Gene im Erbgut wildlebender Tierpopulationen verbreiten kann. Gene Drives erzwingen die Vererbung von neu eingeführten Genen an sämtliche Nachkommen, auch wenn dies die Überlebenschancen der betroffenen Art senkt. Im äußersten Fall könnte die Gene Drive Technologie eine ganze Art in die Ausrottung treiben oder wildlebende Populationen durch gentechnisch veränderte Organismen ersetzen.
Fortpflanzung der ‚Egoisten‘
Die Evolution ist ein langsamer Prozess: Es dauert viele Generationen, bevor sich Veränderungen in der Natur durchsetzen. Das liegt auch an der geschlechtlichen Fortpflanzung, die das Erbgut in jeder Generation neu kombiniert. Neue Eigenschaften stehen in ständiger Konkurrenz zu älteren. An die Nachkommen wird jedoch nur eine von beiden weitergereicht. Welche, das bestimmt der Zufall. Gemäß den Mendelschen Regeln liegt die Wahrscheinlichkeit, dass eine neue Eigenschaft an die Nachkommen vererbt wird, bei 50 Prozent. Eine höhere Vererbungsrate ergibt sich in der Regel nur dann, wenn mit den Eigenschaften Vorteile für das Überleben der Art einhergehen.
Doch nicht alle natürlichen Genanlagen folgen diesen Mendelschen Vererbungsregeln. Bei Pflanzen, Tieren und Menschen gibt es genetische Elemente, die sich mithilfe von Enzymen in andere Teile des Erbguts kopieren, sich selbstständig ausbreiten und damit die Häufigkeit ihrer Vererbung erhöhen. Sie werden häufig als natürlich vorkommende Gene Drives bezeichnet.
Springende Gene
Beispiele sind sogenannte ‚springende Gene‘ (Transposone). ‚Egoistisch‘ werden diese Gene deshalb genannt, weil sie sich im Erbgut ausbreiten können, ohne dass sie der Art nützen. Im Laufe der Evolution haben Pflanzen, Tiere und Menschen einen Umgang mit diesen genetischen Elementen gefunden: Aus einigen entstanden wichtige funktionale, meist regulatorische Einheiten. In vielen anderen Fällen wurden Mechanismen entwickelt, um die ‚springenden Gene‘ im Erbgut stillzulegen (Mehr dazu siehe Infobox).
Gene Drives
Auf einem ähnlichen Prinzip beruhen die sogenannten Gene Drives. Der britische Forscher Austin Burt formulierte im Jahr 2003 die Idee, dass Gene sich rasch ausbreiten können, wenn sie konkurrierende Varianten überschreiben.1 Der natürliche Evolutionsprozess greift dann nicht mehr: Mit Gene Drives kann der Mensch das Erbgut wilder, freilebender Organismen verändern und neue Eigenschaften verbreiten, die einzig seinen Zwecken dienen.
Sogenannte ‚egoistische‘ genetische Elemente finden sich im Erbgut fast aller Lebewesen. Ihre Vermehrung scheint einem Selbstzweck zu folgen. Sie spielen im Rahmen der langen Zeiträume der Evolution aber eine wichtige Rolle. Sie tragen zur Entstehung neuer Genvarianten bei und können unter Umständen wohl auch die Anpassung an veränderte Umweltbedingungen erleichtern. Zahlreiche Schutzmechanismen schränken die unkontrollierte Vermehrung dieser Elemente im Erbgut ein und begrenzen den Schaden für das Lebewesen.
Transposone gehören zu den häufigsten ‚egoistischen‘ Elementen.7 Sie bestehen im Wesentlichen nur aus einem Enzym, das Kopien des Transposons erstellt und diese an anderer Stelle im Erbgut wieder einfügt. Daher stammt auch die Bezeichnung ‚springende Gene‘. Sie wurden ursprünglich von Barbara McClintock entdeckt, die dafür im Jahr 1983 den Nobelpreis erhielt.
Bei Bakterien wurde eine bestimmte Variante von ‚egoistischen‘ Elementen entdeckt, die ,homing‘ Endonukleasen genannt werden.8 Auch sie bestehen nur aus einem einzigen Enzym und können sich selbst zielgenau in bestimmte DNA-Sequenzen einsetzen. Synthetische ‚homing‘ Gene Drives auf Basis von CRISPR/Cas9 wurden nach ihrem Vorbild konzipiert.
Gentechnisch konstruierte, synthetische Gene Drives sind hingegen künstliche genetische Elemente, die mit bestimmten, vom Menschen vorgegebenen Zwecken und Funktionen einhergehen. Sie sind nicht durch evolutionäre Prozesse entstanden und angepasst. Sie sind nicht ‚egoistisch‘, sondern dienen menschlichen Interessen. Evolutionär etablierte Mechanismen, die die Ausbreitung der ‚springenden Gene‘ kontrollieren, sind hier oft unwirksam. Synthetische Gene Drives setzen so eine „mutagene Kettenreaktion“ 9 in Gang, deren Folgen kaum kontrollierbar sind.
In einigen Publikationen wird von Wolbachia Bakterien als ‚natürliche‘ Gene Drives gesprochen. Das ist nicht ganz richtig: Bei Wolbachia handelt es sich um eine über Generationen vererbbare bakterielle Infektion von Insekten.10 Wolbachia Bakterien kommen natürlicherweise in den Zellen bestimmter Insekten, z. B. Fruchtfliegen vor. Sie verringern die Fortpflanzungsfähigkeit der infizierten Insekten. Deshalb wurden mit der Hoffnung der Bekämpfung des Dengue-Fiebers Mücken der Art Aedes aegypti im Labor mit Wolbachia Bakterien infiziert. Dabei stellte man fest, dass bestimmte Wolbachia Bakterien die Übertragung des Dengue-Fiebers auf Menschen blockieren können.11
Feldversuche mit Wolbachia infizierten Mücken fanden erstmals im Jahr 2011 zu Testzwecken in Australien statt.12 Im Unterschied zu synthetischen Gene Drives kommt bei diesem Ansatz keine Gentechnik zum Einsatz. Das bedeutet, dass die mit Gentechnik assoziierten Risiken genetischer Nebeneffekte durch Kreuzung und Interaktion mit wildlebenden Populationen bei Wolbachia Interventionen nicht relevant sind.
Einsatz von Gene Drives in freier Natur
Bislang fanden alle Experimente mit gentechnisch konstruierten Gene Drives ausschließlich im Labor oder in geschlossenen Käfigen statt. Doch eigentlich sind Gene Drives für den Einsatz in der freien Natur gedacht. Sie sollen neue Gene in das Erbgut wildlebender Populationen einführen, auch wenn diese die Überlebenschancen der betroffenen Tierart senken. Ziel ihres Einsatzes in der Natur kann sein, die gesamte Wildpopulation durch gentechnisch veränderte Gene Drive Organismen zu ersetzen oder sie zu dezimieren. Im äußersten Fall könnte ein Einsatz die ganze Art in die Ausrottung treiben.
Erste Freilandversuche mit Gene Drive Mücken könnten bereits im Jahr 2024 in Burkina Faso durchgeführt werden.17 Das wäre ein Versuch ohne jede Absicherung: Mechanismen, die einen Gene Drive in der Natur wirksam kontrollieren, existieren bislang nur in der Theorie.
Nach heutigem Stand der Wissenschaft wäre der Ausgang des Experiments nicht mehr vom Menschen zu kontrollieren. Alle Manipulationen dieser Art an Tieren, Pflanzen sowie an ganzen Ökosystemen wären unumkehrbar.
Seit einigen Jahren finden zu Forschungszwecken auch Freisetzungsversuche mit gentechnisch manipulierten Insekten in der Umwelt statt. Beispielsweise hat die Firma Oxitec in Brasilien seit dem Jahr 2011 mehrfach im Labor gentechnisch veränderten Stechmücken der Art Aedes aegypti freigesetzt, deren gentechnische Veränderung die Mücken oder ihre Nachkommen fortpflanzungsunfähig machen sollte.¹³ Ziel dieser Freisetzungen war eine deutliche Dezimierung der Tropenkrankheiten übertragenden Mückenpopulation. Ob das Ziel erreicht wurde, ist umstritten.¹⁴ In jedem Fall handelte es sich bei keiner der vergangenen Freisetzungen um Insekten, die Gene Drives vererben.
Doch wo liegt der Unterschied zwischen gentechnisch veränderten Organismen (GVO) und gentechnisch veränderten Organismen, die einen Gene Drive (GDO) vererben?
Die neue Dimension der gentechnischen Veränderung von Wildpopulationen mit Gene Drives steht im starken Kontrast zu den bisherigen Zielen, Strategien und Möglichkeiten der Gentechnik:
Bislang sollten gentechnisch veränderte Organismen entweder keine lebensfähigen Nachkommen erzeugen, nicht lange in der Wildnis überleben können oder sie wurden daran gehindert, sich mit wilden Artgenossen zu paaren. Die Anwendung von GVO sollte also außerhalb ihres Entstehungsortes im Labor räumlich oder zeitlich begrenzt bleiben. Diese gentechnisch veränderten Organismen sollten genauso wenig wie ihre veränderten Gene in der Natur überdauern.
Mit diesen Überlegungen bricht der Gene Drive Ansatz radikal. Gentechnisch veränderte Organismen, die Gene Drives vererben, haben im Unterschied zu herkömmlichen GVO zum Ziel, im Labor synthetisierte Gene in Wildpopulationen zu verbreiten oder natürliche Gene auszuschalten. Und das auch, wenn dies der Art schadet oder ihr keinen Überlebensvorteil bietet. Normalerweise würden sich diese Gene im Rahmen der natürlichen Selektion nicht durchsetzen.
Gene Drives verlagern den Ort der gentechnischen Veränderung vom Gentechniklabor in die Natur: Im Fall von CRISPR/Cas9 basierten ‚homing‘ Gene Drives kopiert sich der gentechnische Mechanismus (CRISPR/Cas9) bei jeder Fortpflanzung eines GDO selbstständig ins Erbgut der wildlebenden Nachkommen – über Generationen hinweg. Die durch den Gene Drive ausgelöste ‚erzwungene‘ Vererbung auch schädlicher Gene löst eine theoretisch nicht mehr zu stoppende „mutagene Kettenreaktion“¹⁵ aus.
Durch einen Gene Drive können sich somit vom Menschen verursachte gentechnische Veränderungen viel schneller in Wildpopulationen ausbreiten als es herkömmlichen GVO auf Grundlage der natürlichen Selektionsmechanismen möglich wäre.¹⁶
CRISPR/Cas9 macht’s möglich
Die Verwirklichung von Burts Idee, egoistische genetische Elemente für menschliche Zwecke umzufunktionieren, scheiterte lange Zeit an technischen Hürden. Das änderte sich im Jahr 2012, als die Wissenschaftlerinnen und heutige Nobelpreisträgerinnen Jennifer Doudna und Emanuelle Charpentier das Potential des CRISPR/Cas9 Systems für die Biotechnologie erkannten.2 In Bakterien dient es als eine Art Immunsystem zum Schutz vor Viren: Die CRISPR Sequenz im Erbgut der Bakterien erkennt den Eindringling und aktiviert Enzyme, die das Virus angreifen und sein Erbgut zerschneiden.
Die Forscherinnen erkannten als erste, dass die Kombination aus CRISPR Sequenzen und Cas9 sich dazu nutzen lässt, das Erbgut vieler Lebewesen gezielt zu verändern und neue Abschnitte in deren DNA einzuschleusen. Es war das fehlende Werkzeug, um Burts Idee in die Tat umzusetzen.3 2015 wurde erstmals ein funktionsfähiger CRISPR/Cas9 Gene Drive bei Taufliegen veröffentlicht.4 In den folgenden Jahren waren auch Versuche an Mücken5 und Mäusen6 erfolgreich. Forscher*innen vermuten nun, dass fast jede Tierart mit einem Gene Drive manipuliert werden könnte.
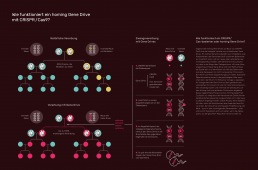
Wie funktioniert ein CRISPR/Cas basierter oder homing Gene Drive?
Sogenannte ,homing‘ Gene Drives auf Basis von CRISPR/Cas9 sind die häufigste Variante von synthetischen Gene Drives. Ein solcher Gene Drive besteht aus mindestens zwei Komponenten: der Genschere Cas9 und einem Botenmolekül. Zusätzlich kann noch ein neues oder verändertes Gen mit eingeschleust werden. Der Gene Drive wird zunächst im Labor in das Erbgut des Zielorganismus, z. B. einer Maus eingeschleust. Dieser Gene Drive wird nach Befruchtung der Eizelle aktiv und identifiziert mit Hilfe des Botenmoleküls eine Zielsequenz im nicht manipulierten Chromosom. Dort führt Cas9 einen Doppelstrangbruch herbei. Natürliche Reparaturmechanismen in der geschädigten Zelle versuchen dann, den Bruch mithilfe einer Vorlage zu reparieren.
Als Vorlage dient der Gene Drive auf dem gentechnisch veränderten Chromosom: Er wird mit hoher Wahrscheinlichkeit vollständig kopiert und innerhalb der Zielsequenz auf dem bislang nicht manipulierten Chromosom eingebaut. Dieser zielgerichtete Prozess wird als ,homing‘ bezeichnet. Zusätzlich zur Integration der Genschere am Zielort können hierbei bestehende Gensequenzen ausgeschaltet und/oder neue zusätzlich eingefügt werden. Dieser Prozess führt letztendlich dazu, dass alle Nachkommen eine Kopie des Gene Drive erben. Der Gene Drive wird bei jeder Fortpflanzung aufs Neue aktiv. Dabei auch in allen nachfolgenden Generationen. Er kommt theoretisch erst zum Halten, wenn die Zielsequenz aus der gesamten Population verschwunden ist.
Quellen
1 Burt A (2003). Site-specific selfish genes as tools for the control and genetic engineering of natural populations. Proc Biol Sci 270:921
2 Werren JH (2011). Selfish genetic elements, genetic conflict, and evolutionary innovation. Proc Natl Acad Sci U S A. 108 Suppl 2:10863
3 Stoddard BL (2011). Homing endonucleases: from microbial genetic invaders to reagents for targeted DNA modification. Structure 19:7
4 Gantz VM, Bier E (2015). The mutagenic chain reaction: a method for converting heterozygous to homozygous mutations. Science 348:442
5 O’Neill SL, Giordano R, Colbert AM, Karr TL, Robertson HM (1992). 16S rRNA phylogenetic analysis of the bacterial endosymbionts associated with cytoplasmic incompatibility in insects. Proc Natl Acad Sci U S A. 89(7):2699–702
6 Moreira LA, Iturbe-Ormaetxe I, Jeffery JA, Lu G, Pyke AT, Hedges LM, Rocha BC, Hall-Mendelin S, Day A, Riegler M, Hugo LE, Johnson KN, Kay BH, McGraw EA, van den Hurk AF, Ryan PA, O’Neill SL (2009). A Wolbachia symbiont in Aedes aegypti limits infection with dengue, Chikungunya, and Plasmodium. Cell.139(7):1268–78
7 World Mosquito Website (2020). About us. Our story. Online: https://www.worldmosquitoprogram.org/en/about-us/our-story [letzter Zugriff: 22.10.2020]
8 Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E (2012). A Programmable Dual-RNA-Guided DNA Endonuclease in Adaptive Bacterial Immunity. Science 337:816
9 Esvelt KM, Smidler AL, Catteruccia F, Church GM (2014). Concerning RNA-guided gene drives for the alteration of wild populations. Elife 17:e03401
10 Gantz VM, Bier E (2015). The mutagenic chain reaction: a method for converting heterozygous to homozygous mutations. Science 348:442
11 Kyrou K, Hammond AM, Galizi R, Kranjc N, Burt A, Beaghton AK, Nolan T, Crisanti A (2018). A CRISPR-Cas9 gene drive targeting doublesex causes complete population suppression in caged Anopheles gambiae mosquitoes. Nat Biotechnol 36:1062
12 Grunwald HA, Gantz VM, Poplawski G, Xu XS, Bier E, Cooper KL (2019). Super-Mendelian inheritance mediated by CRISPR-Cas9 in the female mouse germline. Nature 566:105
13 Van Woensel L, Van Steerteghem J (Scientific Foresight Unit (STOA), European Parliamentary Research ServiceScientific Foresight Unit (EPRS, 2019). The Science and ethics of gene drive technology. Case Study: Eradicating malaria. Working Breakfast; 2019 Mar 19; Europäisches Parlament, Brüssel, Belgien. p. 6. Online: https://www.europarl.europa.eu/cmsdata/161962/1-Booklet.pdf [letzter Zugriff: 22.10.2020]
14 Oxitec Website (2020). Oxitec. Brazil. Abingdon. Online: https://www.oxitec.com/brazil [letzter Zugriff: 22.10.2020]
15 Wallace H, Jackson A, Li Ching L, Sirinathsinghji E, Mayet M (2019). Oxitec’s failed GM mosquito releases worldwide: Forewarnings for Africa and the Target Malaria project. Online: acbio.org.za/sites/default/files/documents/Oxitec_failed_GM_mosquito_releases_worldwide [letzter Zugriff: 22.10.2020]
16 Gantz VM, Bier E (2015). The mutagenic chain reaction: a method for converting heterozygous to homozygous mutations. Science 348:442
17 Simon S, Otto M, Engelhard M (2018). Synthetic gene drive: between continuity and novelty: Crucial differences between gene drive and genetically modified organisms require an adapted risk assessment for their use. EMBO Rep 19 (5)